| 2014年8月29日 |
| 独立行政法人 日本原子力研究開発機構J-PARCセンター |
| DNAの曲がりやすさにも遺伝子発現情報が含まれている |
| - J-PARCにおける中性子準弾性散乱実験とシミュレーション計算により、DNAと水和水の運動の観測に成功 - |
| 【 発表のポイント 】 ○ J-PARCにおける中性子準弾性散乱実験とシミュレーション計算により、DNA構造の曲がりやすさが塩基配列によって異なることを実証した。
○ DNAの曲がりやすさが水和水の運動と密接に関係していることを突き止めた。 |
| 独立行政法人日本原子力研究開発機構 (理事長 松浦祥次郎。以下「原子力機構」という。) 原子力科学研究部門量子ビーム応用研究センターの分子ダイナミクス研究グループ中川洋研究副主幹と分子シミュレーション研究グループ米谷佳晃研究副主幹は、中性子準弾性散乱実験1) とコンピュータシミュレーションにより、塩基配列2) によってDNAの曲がりやすさが異なることを実証するとともに、DNAの二重らせん構造の副溝3) にある水和水4) の運動とも密接に関係していることを明らかにしました。 これまでの研究では、DNAの曲がりやすさの程度は、塩基配列によって異なることが分かっていました。
今回の研究では、シミュレーション計算で曲がりやすさが大きく異なると予測された二つのDNAに関して、DNAの水和水の運動を直接観測できる中性子準弾性散乱実験をJ-PARCで実施し、その運動の詳細をシミュレーション計算により追跡した結果、DNAの曲がりやすさは塩基配列によって異なることを実証しました。さらに、実験データとシミュレーションデータを統合して解析することによって、1) 運動の大きいDNAの方が曲がりやすいこと、2) DNAの水和水の運動は水素結合の寿命と高い相関を示すことを見出しました。つまり、DNAの曲がりやすさはDNA二重らせんの副溝に存在する水和水の水素結合の寿命によって決まるという分子メカニズムを明らかにしました。
本成果は、DNA配列には塩基の情報だけではなく、曲がりやすさという情報も含んでいることを実証したものです。今回実証したDNAの情報は、DNA配列から特定の遺伝子を働かせたり抑制したりする仕組みの解明に貢献できると期待されます。
本研究成果は、2014年8月29日に「Physical Review E」に掲載される予定です。 |
| 【 本件に関する問合せ先 】 (研究内容について) 原子力科学研究部門 量子ビーム応用研究センター 量子ビーム機能性分子解析技術研究ユニット 分子シミュレーション研究グループ グループリーダー 河野 秀俊 TEL:0774-71-3465, FAX:0774-71-3460
(報道担当) 広報部報道課長 中野 裕範 TEL:03-3592-2346, FAX:03-5157-1950 |
| |
| 【 研究開発の背景と目的 】 多くの生物種で生命の設計図であるゲノム (DNA配列) が明らかにされている現在の生物学では、ゲノムから生命機能を解析・予測する研究基盤が整ってきました。しかし、遺伝子発現の初期過程のDNAから mRNA5) に情報が読み出されるプロセスなどDNAの動態については未解明な点が残されています。特に、読み出しの際のたんぱく質とDNAの複合体におけるアミノ酸と塩基の相互作用には明確な配列の対応関係が見られないことが、ゲノムから機能を予測することを困難にしていました。最近の研究では、" 直接認識 "6) とよばれるたんぱく質とDNAの直接的な相互作用だけでなく、" 間接認識 "6) とよばれる、水和水を介した相互作用やDNAの配列依存的な構造の変形のしやすさ (図1) が、ゲノムにコードされている情報として重要であることが分かってきました (Sarai, A. & Kono, H. (2005) Protein-DNA Recognition Patterns and Predictions, Annu Rev Biophys Biomol Struct 34, 379-398) 。 この間接認識では、配列依存的なDNAの変形のしやすさや水和状態を解析することが重要であり、DNAの運動や水和との関係性を、ダイナミクスを直接的に観測できる実験的手法で実証することが求められていました。中性子準弾性散乱実験ではDNA分子の運動を観測できるので、本研究では、中性子準弾性散乱実験とコンピュータシミュレーションを組み合わせて解析することで、配列によって異なるDNAの運動と水和の関係の解明を目的としました。 |
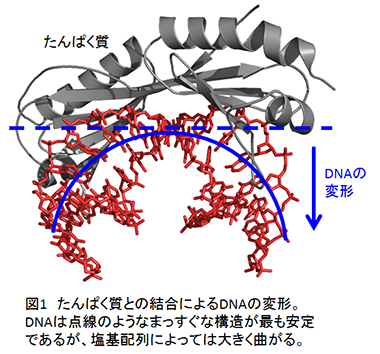  *クリックすると、大きく表示されます。 *クリックすると、大きく表示されます。 |
| |
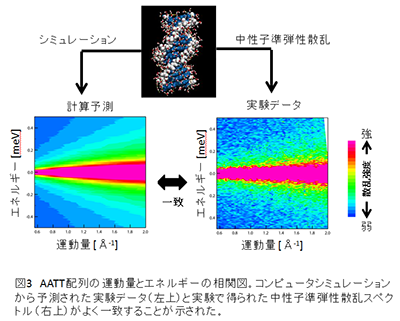
| 【 研究の手法と成果 】 本研究では、これまで行ってきた系統的なコンピュータシミュレーション計算結果の解析で、最も硬いと予測されている5'CGCGAATTCGCG3'と最も柔らかいと予測されている5'CGCGTTAACGCG3'の二種類のDNA配列を研究対象とし、DNAと水和水のシミュレーション計算と、J-PARCの物質・生命科学実験施設 (MLF) 7) のAMATERAS装置を用いて中性子準弾性散乱実験を実施しました (図2) 。シミュレーションでは、DNAと水和水のすべての原子の時間的な変化を追跡することができますが、実際に現実の世界で起こっている時間スケールの現象を正確に再現できているかどうかの確証はありません。一方で、中性子準弾性散乱実験では、すべての原子をつぶさに観測はできませんが、平均像として実際に起こっていることが分かります。そこで、この二つの研究方法の長所を活かし、欠点を補うことで、DNAや水和水の運動を正確に解析することが可能となります。図3に示すように、運動量とエネルギーの相関図は、計算と実験とで良く対応していることが確認できました。さらに、中性子準弾性散乱実験データのより詳細な解析を行った結果、DNAの構造変化のしやすさの指標となる原子の移動距離はAATTよりもTTAAが長いことが分かり、シミュレーション計算で変形のしやすさが異なると予測された二つのDNAの運動の違いの妥当性が実証されました。 |
  *クリックすると、大きく表示されます。 *クリックすると、大きく表示されます。 |
| |
| 次にこのDNA構造の変形のしやすさの原因を調べるために、シミュレーションデータから水和状態の詳細な解析を行いました。その結果、副溝の水分子とDNAとの水素結合の寿命が、AATTの方がTTAAに比べて長いことが分かりました (図4) 。これはAATTの硬いDNAの方が、DNAと水和水の間の水素結合の組み換えに時間がかかり、水和水の運動も遅いことを示しています。このように、DNAの変形しやすさのメカニズムを分子レベルで明らかにすることができました。 |
| |
  *クリックすると、大きく表示されます。 *クリックすると、大きく表示されます。 |
| |
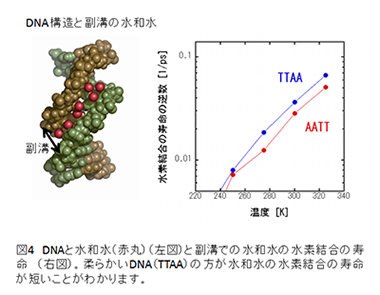  *クリックすると、大きく表示されます。 *クリックすると、大きく表示されます。 |
| |
| 【 今後の期待 】 本研究のポイントは、DNAがもつ本来の構造特性を解明するものであり、配列によってその特性が異なることを示すだけでなく、それが水和水の運動と密接に関係していることを示した点にあります。従来、DNAの認識機構を説明するとき、塩基配列とたんぱく質のアミノ酸の直接的な相互作用が着目されてきましたが、本研究はDNA構造の変形しやすさという間接的な相互作用という視点の重要性を示すものです。今回は二つのDNA配列について比較しましたが、今後、様々な配列について系統的に調べていくことで、例えば新たなDNA結合たんぱく質の分子設計などが可能になってくると期待できます。
また本研究は、中性子準弾性散乱実験とコンピュータシミュレーションを組み合わせた研究としても特色があります。実験と計算を相補的・相乗的に融合させた本研究の解析は、単に互いの手法の欠点を補い合うという意味だけでなく、計算により実験情報から埋もれた情報を取り出すという意味でも有効です。今後も、J-PARC/MLFとコンピュータシミュレーションの融合解析により、DNAやたんぱく質など生命活動の根幹として働いている分子の機能発現の仕組みが明らかになってくると期待されます。 |
| |
|
©2014 J-PARC Center. All rights reserved. |

